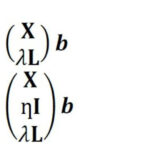- Startseite
- Forschungsabteilungen
- Photonic Data Science
- Forschungsergebnisse
- Untersuchungen zur Raman-Spektraldatenanalyse
Untersuchungen zur Raman-Spektraldatenanalyse
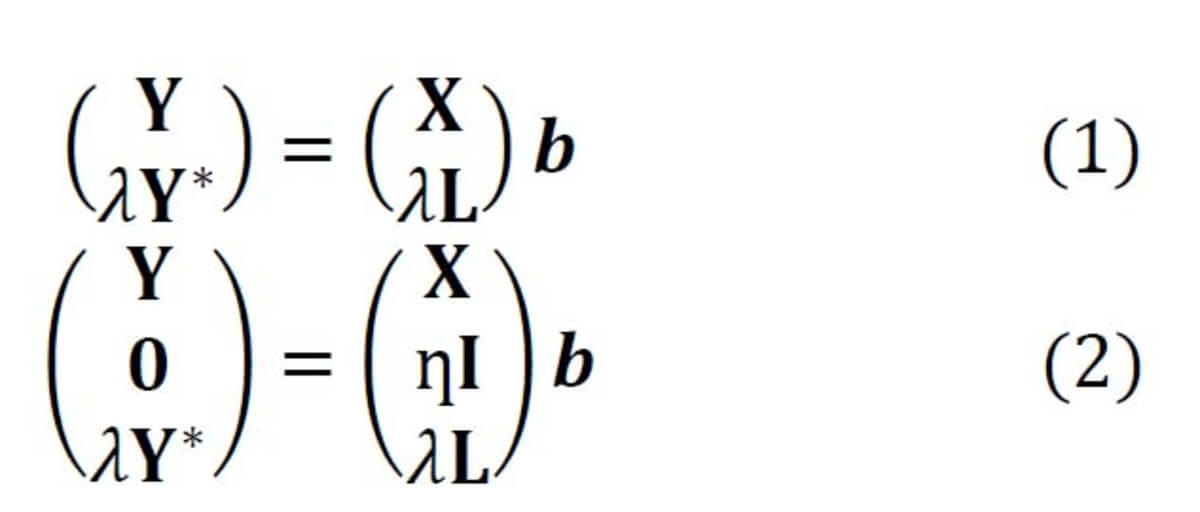
23.04.2018
Wenn ein auf Raman-Spektroskopie basierendes Klassifikationsmodell verwendet wird, um Daten vorherzusagen, die, verglichen mit den Trainingsdaten, auf einem anderen Gerät oder an einem anderen biologischen Replikat gemessen wurden, ist die Leistungsfähigkeit des Modells in der Regel gering. Dieses Problem kann durch Transfermethoden angegangen werden. Wir untersuchten Transfermethoden auf Basis der Tichonow-Regularisierung (TR) und überprüften die Transferleistung des Modells.
Von Shuxia Guo // Ralf Heinke // Stephan Stöckel // Petra Rösch // Jürgen Popp // Thomas Bocklitz
Die Raman-Spektroskopie hat sich als vielseitiges Werkzeug für biologische Studien wie Toxikologie, Mikrobiologie, Wirkstoffforschung, Stoffwechseluntersuchungen und forensische Analysen bewährt [1]. Diese Anwendungen der Raman-Spektroskopie sind nur durch die Verwendung der Chemometrie bei der Raman-Spektraldatenanalyse möglich. Die Chemometrie verbessert die Empfindlichkeit von Raman-basierten biologischen Nachweisen, da sie in der Lage ist, durch biologische Veränderungen hervorgerufene subtile spektrale Variationen zu unterscheiden. Deshalb wenden wir die Chemometrie in Raman-basierten biologischen Studien an und untersuchen die Möglichkeiten, Ergebnisse der chemometrischen Modelle zu verbessern. Eines unserer untersuchten Themen ist das Problem des Modelltransfers. Dies tritt in Erscheinung, wenn Testdaten signifikante Unterschiede zu den Trainingsdaten aufweisen, die auf Variationen zwischen verschiedenen Replikaten bzw. Instrumenten zurückzuführen sind. Modelltransferansätze ermöglichen es dem trainierten Modell, diese neuen Daten erfolgreich vorherzusagen, ohne eine große Anzahl neuer Trainingsproben zu verwenden, die unter den veränderten Bedingungen vermessen wurden. Dies ist vor allem dann notwendig, wenn neue Trainingsmuster teuer oder gar nicht zu beschaffen sind, wie es in den meisten biologischen Studien der Fall ist. Die Trainingsdaten werden als Primärdaten bezeichnet, während die neuen Daten, die unter verschiedenen Bedingungen gemessen werden, als Sekundärdaten bezeichnet werden.
Die gebräuchlichste Modelltransfermethode in der Raman-Spektroskopie ist die Standardkalibrierung [2], bei der die spektralen Variationen zwischen Instrumenten auf Grundlage von standardisierten Messungen eliminiert werden. Die Standardkalibrierung hat jedoch keinen Einfluss auf die Variationen zwischen verschiedenen Replikaten, was eine der Hauptursachen für eine fehlgeschlagene Vorhersage in biologischen Anwendungen ist. Aus diesem Grund haben wir zwei Ansätze entwickelt, die auf der Tichonow-Regularisierung (TR) basieren [3], die als TR1und TR2bezeichnet werden [4]. Die Gleichungen dieser beiden Ansätze sind jeweils in Gl. (1-2) angegeben. Die Methode basiert auf der Ergänzung der (primären) Trainingsdaten (X,Y) mit einigen sekundären Trainingsproben (L,Y*), welche als Transferspektren bezeichnet werden.
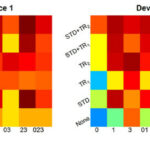
Die Untersuchung basiert auf Raman-Spektren von drei Sporenarten (B. mycoides, B. subtilis und B. thuringiensis), die an vier Geräten gemessen wurden. Ein Drei-Gruppen-Klassifizierungsmodell wurde mittels partieller Regression der kleinsten Quadrate konstruiert. Jedes Gerät wurde einmal für Sekundärdaten verwendet und von Modellen vorhergesagt, die mit einem oder mehreren der anderen Geräte trainiert wurden. Insbesondere wurden für jeden Fall von Sekundärdaten zufällig fünfzehn Sekundärspektren als Optimierungsspektren ausgewählt, von denen jeweils fünf zu einer Gruppe gehörten. Die Parameter λ und ƞ wurden durch genetische Algorithmen optimiert, um die Vorhersage dieser Optimierungsspektren zu maximieren. Die Berechnung wurde auf Raman-Spektren mit oder ohne Standardkalibrierung (STD) durchgeführt. Die Vorhersagegenauigkeit der Sekundärdaten wurde in Abbildung 1 dargestellt. Die Genauigkeit nach Modelltransfer im Vergleich zur Vorhersage ohne Modelltransfer wurde deutlich verbessert. Beide Methoden, TR1und TR2, konnten die Vorhersage deutlich verbessern, auch wenn keine Standardkalibrierung durchgeführt wurde. Trotzdem ist TR2TR1überlegen.
Gefördert von: China scholarship council, BMBF, EU