- Startseite
- Forschungsabteilungen
- Biophysikalische Bildgebung
- Forschungsergebnisse
- Biophysikalische Bildgebung mit hoher räumlicher und zeitlicher Auflösung
Biophysikalische Bildgebung mit hoher räumlicher und zeitlicher Auflösung
28.03.2019
Die Fluoreszenzmikroskopie ist ein leistungsfähiges Werkzeug zur Beobachtung zellulärer Strukturen über Raum und Zeit. Allerdings ist die Auflösung für Strukturen kleiner 200 nm in der Regel zu begrenzt und die Dynamik liegt nur im Sekunden-Bereich. Wir stellen hier die Entwicklung der Beam-Scanning-Fluoreszenzkorrelationsspektroskopie in Kombination mit hochauflösender STED-Mikroskopie vor, was deutlich höhere Auflösungen ermöglicht, beispielsweise zur Bestimmung der Diffusionsdynamik von Lipidmembranen.
Von Christian Eggeling
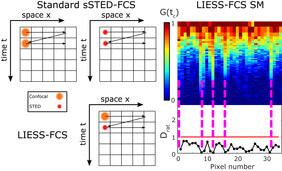
Die zelluläre Signalübertragung hängt stark von der Diffusions- und Interaktionsdynamik in der zellulären Plasmamembran ab, dem „Tor“ zur Zelle. Diese Dynamik wird durch laterale Heterogenität in der Organisation der molekularen Plasmamembran moduliert. Diese Heterogenität und die zugrundeliegenden Strukturen bzw. molekularen Interaktionsdynamiken können durch Erforschung der molekularen Diffusionseigenschaften in der Plasmamembran über Raum und Zeit untersucht werden. Ein weit verbreiteter Ansatz zur Erforschung der molekularen Diffusion auf Ebene der zellulären Plasmamembran ist die Fluoreszenzkorrelationsspektroskopie (FCS). FCS wird üblicherweise verwendet, um die durchschnittlichen Transitzeiten (τD) von Molekülen durch das Beobachtungsvolumen eines Konfokalmikroskops zu bestimmen, um Werte der Diffusionskoeffizienten (D) zu erhalten, die Veränderungen in der molekularen Diffusion aufzeigen, verursacht beispielsweise durch Änderungen der Membranviskosität oder molekulare Wechselwirkungen. Darüber hinaus ermöglicht FCS die Untersuchung der nicht durch brownsche Bewegung behinderten Diffusion, die durch molekulare Wechselwirkungen und Einschlüsse verursacht wird und durch die Diffusionsmoden der Moleküle offenbart wird. Solche Moden können durch die Aufzeichnung von FCS-Daten für Beobachtungspunkte unterschiedlicher Größe gemessen werden, wodurch die Abhängigkeit von τDund D vom Durchmesser d der Beobachtungspunkte bestimmt werden kann. Von τD(d) (bzw. D(d)) lässt sich beispielsweise zwischen freier (brownscher) Diffusion und transientem Trapping in sich langsam bewegenden oder immobilisierten Entitäten (Trapped Diffusion) unterscheiden. Leider können Parameter wie Verweildauer oder Größe der Trapping-Plätze mit einem herkömmlichen Konfokalmikroskop nur extrapoliert werden, da die relevanten molekularen Skalen unterhalb der Beugungsbegrenzung (d~240 nm) liegen. Abhilfe schafft hier die Realisierung von FCS-Aufnahmen mit Beobachtungspunkten unterhalb der Beugungsbegrenzung (z.B. von d~240 nm bis d < 50 nm), wie sie durch die hochauflösende STED-Mikroskopie erzeugt werden. Um die räumliche Heterogenität und die damit verbundenen räumlichen Diffusionsmoden genau zu verstehen, müssen FCS-Daten an verschiedenen Stellen gleichzeitig aufgezeichnet werden, wie bei Scanning-STED-FCS (sSTED-FCS), wo mehrere FCS-Daten gleichzeitig für jedes Pixel entlang einer schnell gescannten Zeile aufgezeichnet werden. Leider war sSTED-FCS bisher nicht in der Lage, Diffusionsmoden genau zu charakterisieren, da Werte für τD(bzw. D) nur für jeweils einen Beobachtungspunktdurchmesser d bestimmt werden konnten. Hierfür haben wir die zeilenüberlappende Anregungs-Scanning-STED-FCS (LIESS-FCS) entwickelt, die durch schnelles Beam-Scanning entlang einer Zeile mit alternierender Laserbeleuchtung für jedes Pixel die offenkundigen Diffusionskoeffizienten für zwei verschiedene Beobachtungspunktgrößen liefert, von denen einer den beugungsbegrenzten Konfokal- und der andere den hochaufgelösten STED-Mikroskopieaufnahmen entspricht. Wir haben unseren LIESS-FCS-Ansatz mit Untersuchungen von nanoskaligen Lipiddiffusionsmoden in der Plasmamembran lebender Zellen validiert und dabei transiente Interaktions- (bzw. Trapping-)Stellen für bestimmte Lipide hervorgehoben.
Genauer gesagt, wird bei sSTED-FCS entweder der größere konfokale (dconf~240 nm) bzw. der kleinere STED (dSTED<<200 nm) Beobachtungspunkt schnell und mehrfach über die Probe entlang einer Zeile (oder eines Kreises) gescannt, wodurch für jedes Zeilenpixel zeitliche Intensitätsdaten erzeugt werden. Die anschließende Berechnung der zeitlichen Korrelationsfunktion für jedes Pixel erzeugt die im Raum zusammengefassten Autokorrelationsfunktionen in den sogenannten Korrelationsteppichen. Die Anpassung jeder Korrelationskurve ergibt Werte für τDund den offenkundigen Diffusionskoeffizienten Dconf = D(dconf) und DSTED = D(dSTED)für konfokale bzw. STED-Aufnahmen. Mit sSTED-FCS können Dconfund DSTEDnur nacheinander und nicht gleichzeitig bestimmt werden. Da sich die Diffusionseigenschaften an den einzelnen Pixeln zwischen konfokalen und STED-Aufnahmen aufgrund der globalen Zelldynamik (z.B. zelluläre Bewegungen, Variationen in der Plasmamembran-Topologie) geändert haben können, lassen sich die Werte von Dconfund DSTEDnicht paaren, um räumlich aufgelöste D(d)-Abhängigkeiten zu bestimmen. Im Gegensatz dazu werden mit LIESS-FCS die konfokalen und STED-basierten Beobachtungspunkte abwechselnd gescannt (d.h. auf zeilenweiser Basis), wodurch Korrelationsteppiche für konfokale und STED-Moden und damit Werte für Dconfund DSTEDfür jedes Pixel quasi gleichzeitig innerhalb derselben Messung erzeugt werden. Dies ermöglicht nun die direkte Offenlegung von Diffusionsmoden für jedes Pixel, indem die Werte von Dconfund DSTEDin Beziehung gesetzt werden, indem das Verhältnis Drat = DSTED/Dconffür jedes Pixel berechnet wird. Die Werte von Dratliefern einzigartige Informationen über die nanoskaligen Diffusionseigenschaften, da sie für verschiedene nanoskalige Diffusionsmoden variieren: Drat = 1 für freie und Drat < 1 für Trapping Diffusion.
Wir verwendeten LIESS-FCS, um die Diffusionseigenschaften von fluoreszierend markiertem Phosphoethanolamin-Lipid (DPPE) im Detail zu untersuchen und mit einem fluoreszierend markierten Sphingomyelin-Lipid (SM) in der Plasmamembran lebender Zellen zu vergleichen. Frühere sSTED-FCS-Experimente haben eine weitgehend freie homogene Diffusion für DPPE und räumlich abgesetzte Stellen einer verlangsamten Diffusion im Falle von SM gezeigt, die nur in den STED-Aufnahmen sichtbar sind. Aufgrund des Fehlens gleichzeitiger Informationen aus konfokalen Aufnahmen konnte diese Beobachtung mit sSTED-FCS jedoch nicht direkt auf Trapping-Interaktionen zurückgeführt werden. Die LIESS-FCS-Modalität ermöglicht es nun, diese Stellen direkt mit Trapping Diffusion in Verbindung zu bringen, da Dratnur an diesen räumlichen Positionen << 1,0 ist, während Dratdazwischen jeweils nahe 1,0 liegt (nahezu freie Diffusion wie mit DPPE kontinuierlich erfasst). Daher ist jede andere auftauchende räumliche Heterogenität, wie sie bereits in den konfokalen Korrelationsteppichen des DPPE zu finden ist, noch immer durch freie Diffusion gekennzeichnet, d.h. sie beziehen sich trotz der offensichtlichen Heterogenität nicht auf Trapping-Wechselwirkungen. Eine mögliche Ursache für diese Heterogenität kann die ungleichmäßige Plasmamembran-Topologie mit Krümmungen sein.
Insgesamt zeigen unsere Daten die Fähigkeit von LIESS-FCS, die räumliche Heterogenität im molekularen Diffusionsverhalten direkt zu beobachten (z.B. räumlich abgesetzte Stellen mit Trapping Diffusion). Damit bietet LIESS-FCS ein einzigartiges Werkzeug zur Untersuchung der lateralen Organisation von Zellmembranen auf variablen Längenskalen, wobei Verzerrungen durch biologische Heterogenität oder Photobleichungsartefakte berücksichtigt und möglicherweise langjährige Fragen der funktionellen Membranheterogenität beantwortet werden.
