- Startseite
- Forschungsabteilungen
- Photonic Data Science
- Forschungsergebnisse
- Merkmalsextraktion bei der Analyse von Raman-Spektraldaten
Merkmalsextraktion bei der Analyse von Raman-Spektraldaten
08.12.2020
Eine auf Raman-Spektroskopie basierende Klassifizierung kann das Muster der Inter-Gruppen-Unterschiede aufgrund der Intra-Gruppen-Spektralvariationen nicht lernen. Eine natürliche Lösung ist eine gut konzipierte Methode zur Merkmalsextraktion, die die Intra-Gruppen-Variationen von den Inter-Gruppen-Unterschieden trennen kann; der Klassifikator wird dann nur auf der Grundlage der letzteren Informationen erstellt. Zu diesem Zweck haben wir zwei häufig angewandte Methoden zur Merkmalsextraktion, die Hauptkomponentenanalyse (PCA) und die partielle kleinste Quadrate (PLS), modifiziert, um die Merkmale zu extrahieren, die lediglich die Unterschiede zwischen den Gruppen darstellen.
Von Shuxia Guo // Petra Rösch // Jürgen Popp // Thomas Bocklitz
Die Raman-Spektroskopie erlebte in den letzten zwei Jahrzehnten ein dramatisches Wachstum in biologischen Anwendungen, einschließlich, aber nicht beschränkt auf Toxikologie und Forensik, Mikrobiologie, Arzneimittelentdeckung, Stoffwechseluntersuchungen und sogar In-vivo-Detektion [1]. Dies hat weitgehend von der Entwicklung der Chemometrie profitiert, bei der die Raman-Signale in hochrangige chemische und/oder biologische Informationen von Interesse übersetzt werden, meist unter Verwendung eines Klassifikationsmodells [2]. Die Raman-basierte Klassifizierung wird jedoch oft durch die spektralen Variationen innerhalb der Gruppe beeinträchtigt, die die subtilen spektralen Unterschiede zwischen den Gruppen, die von Interesse sind, leicht überdecken können. Um dieses Problem in den Griff zu bekommen und damit die Leistung der Klassifikation zu verbessern, haben wir zwei häufig verwendete Methoden zur Merkmalsextraktion, PCA und PLS, so modifiziert, dass die extrahierten Merkmale lediglich die Unterschiede zwischen den Gruppen darstellen. Die Details sind in der Publikation [3] zu finden. Im Folgenden werden die Methoden kurz zusammengefasst, zusammen mit ihren Ergebnissen auf zwei Raman-Spektraldatensätzen, die von biologischen Proben gemessen wurden.
Die Idee der modifizierten PCA ist wie folgt gegeben:
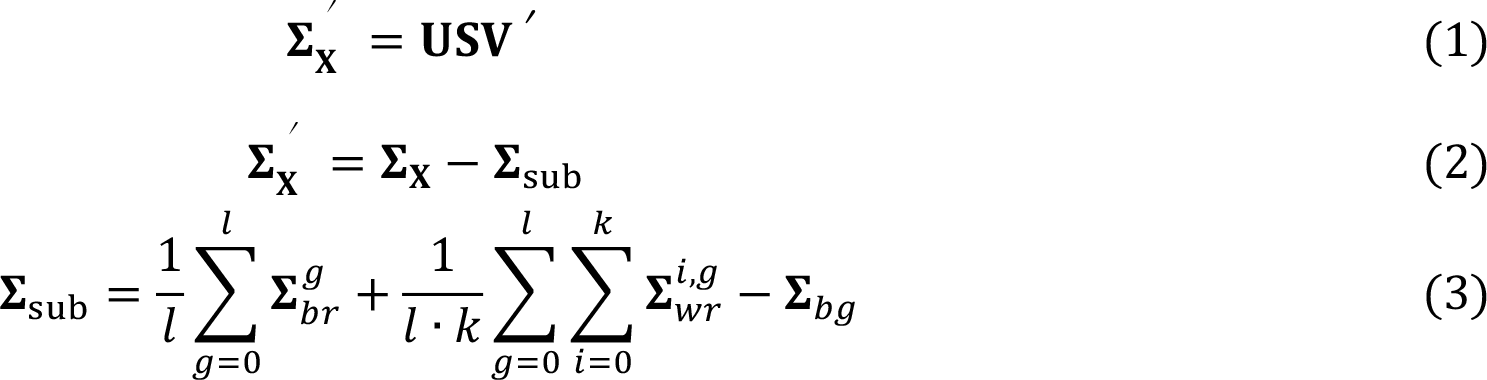
∑x, ∑sub und ∑’x repräsentieren die Gesamtkovarianz, die Intra-Gruppen-Kovarianz bzw. die Inter-Gruppen-Kovarianz. Die ist die Kovarianzmatrix der mittleren Spektren jedes Replikats, das zur g-ten Gruppe gehört, und die ist die Kovarianzmatrix des i-ten Replikats, das zur g-ten Gruppe gehört. ∑bg ist die Kovarianzmatrix der mittleren Spektren der einzelnen Gruppen. Durch Subtraktion von ∑sub von ∑x sollen die resultierenden Hauptkomponenten V aus der Singulärwertzerlegung (Gl. (1)) lediglich die Unterschiede zwischen den Gruppen anzeigen.
Das modifizierte PLS (mPLS) wurde durch Orthogonalisierung der Projektionsvektoren (P) gegen Lsub während der Iteration des SIMPLS-Algorithmus erreicht. Die Orthogonalisierung erfolgte gemäß Gl. (4), wobei Lsub die aus Gl. (1) resultierenden Ladungen sind, die die Intra-Gruppen-Variationen darstellen. Die resultierenden Projektionsvektoren Pot wurden verwendet, um Score-Vektoren von mPLS zu erhalten.
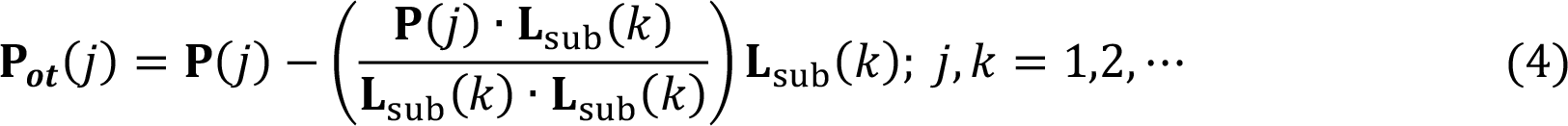
Die Methoden wurden mit zwei Raman-Spektraldatensätzen verifiziert, die von Dickdarmgewebe von Mäusen bzw. von Bakterien gemessen wurden. Die Leistung wurde anhand der mittleren Sensitivität der Klassifikationen mit ihren gewöhnlichen Gegenstücken verglichen. Dementsprechend wurde bewiesen, dass die mPCA die Generalisierung des Klassifikators verbessert, während die mPLS hilft, Overfitting zu vermeiden. Aus Platzgründen werden in den folgenden Abschnitten nur die Ergebnisse der Mäusedaten beschrieben.
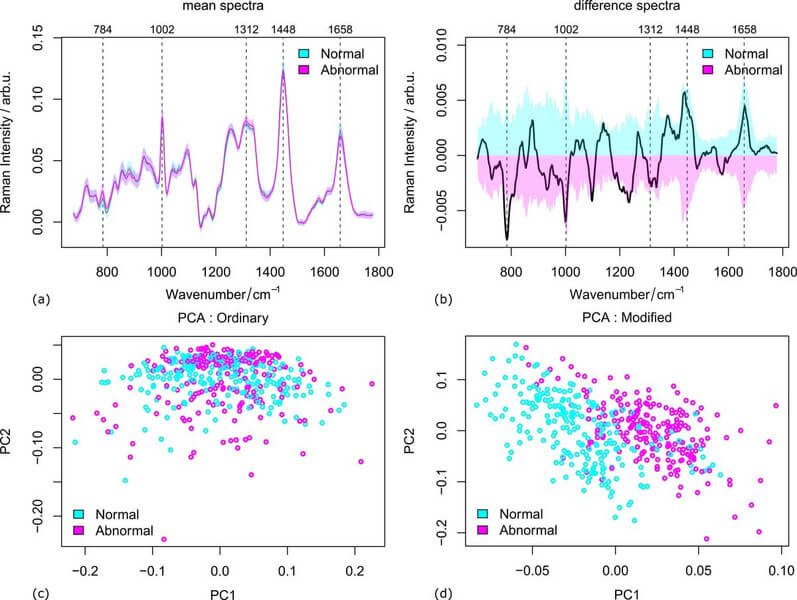
Die Daten der Mäuse wurden in zwei Fällen gemessen: vollständig präparierte Proben von 47 Individuen und Biopsieproben von 97 Individuen (Details siehe ref. [4]). Die mittleren Spektren der beiden abnormalen von den normalen Proben sind in Abbildung 1 (a-b) zusammen mit den Varianzen und ihren Differenzspektren aufgetragen. Die Peaks, bei denen sich die beiden Gruppen signifikant unterscheiden, sind durch vertikale Linien markiert. Aus den Score-Plots in Abbildung 1(c-d) ist ersichtlich, dass die beiden Gruppen mit den ersten beiden Komponenten der gewöhnlichen PCA kaum zu unterscheiden sind. Im Fall der modifizierten PCA wurde eine wesentlich bessere Trennung beobachtet. Das heißt, die Unterschiede zwischen den Gruppen werden durch die modifizierte PCA besser extrahiert.
Zusätzlich haben wir die Leistung der mPCA und mPLS entsprechend der Vorhersage auf Biopsieproben mit dem Klassifikator getestet, der auf vorbereiteten Proben aufgebaut wurde. Die mittlere Empfindlichkeit der Vorhersage ist in Abbildung 2 dargestellt. Obwohl die gewöhnliche und modifizierte PLS eine vergleichbare höchste mittlere Empfindlichkeit über verschiedene Anzahl von Komponenten (nLV) ergab, zeigt sich die Klassifikation im Fall von mPLS stabiler zur Anzahl von Komponenten (nLV). Im Gegensatz dazu war die höchste mittlere Sensitivität mit mPCA deutlich besser als mit seinem normalen Gegenstück. Dies zeigt, dass der Klassifikator im ersten Fall toleranter gegenüber den Unterschieden zwischen den präparierten und den Biopsieproben ist (d. h. eine bessere Generalisierbarkeit der Klassifikation).